الگوریتم هیرشبرگ
در علوم رایانه، الگوریتم هیرشبرگ (به انگلیسی: Hirschberg's Algorithm) الگوریتمی برای همترازسازی بهینهٔ دو توالی است که دان هیرشبرگ (Dan Hirschberg) در سال ۱۹۷۵ آن را ارائه کرد.
در این الگوریتم برای ارزیابی همترازیها از فاصله لوناشتاین استفاده میشود؛ در همترازی بهینه مجموع هزینههای درج، حذف و جایگزینکردن حروف برای یکسانکردن دو رشته، کمینهٔ تمام همترازیهای ممکن است.
الگوریتم نیدلمن-وانچ یکی از اولین الگوریتمهایی است که برای همترازسازی سراسری دو توالی از برنامهنویسی پویا استفاده میکند. الگوریتم هیرشبرگ در واقع نسخهای از الگوریتم نیدلمن-وانچ است که با تقسیم و حل مسئله را حل میکند و مصرف حافظهٔ بهینهتری دارد.
ایدههای اصلی
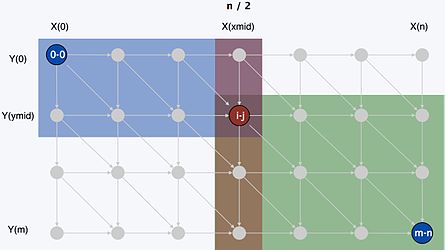
هوشمندی الگوریتم هیرشبرگ در استفاده از مشاهدههای زیر است:
- برای محاسبهٔ امتیاز (یا فاصلهی) بهینهٔ همترازی، کافی است سطر قبلی و سطر جاری را ذخیره کرد (و نه کل سطور ماتریس امتیاز نیدلمن-وانچ).
- اگر
همترازی سراسری بهینهٔ رشتههای X و Y باشد و
تقسیمبندی دلخواهی از X باشد، تقسیمبندی
از Y وجود دارد که
.
این الگوریتم از ایدهٔ Savitch در نظریهٔ پیچیدگی الهام گرفتهاست.
امتیازدهی
برای محاسبهٔ امتیاز همترازی (درج و حذف، جایگزینی و تطابق) از ماتریسهای امتیازدهی استفاده میشود. برای مثال، در ماتریس بلوسام-۶۲, امتیاز جایگزینی آمینواسیدهای Ala و Trp، تطابق آمینواسید Ala و حذف آمینواسید Ala، به ترتیب ۳-، ۴ و ۴- است.
معمولاً برای امتیازدهی همترازیهای آمینواسیدی (توالیهای پروتئینی) از ماتریسهای بلوسام یا ماتریسهای جهش پذیرفتهٔ نقطهای استفاده میشود.
الگوریتم
برای رشتهی مفروض 




برای دو حرف x و y که به ترتیب از رشتههای X و Y اند؛ امتیاز حذف x, درج y و جایگزینی x با y به ترتیب برابر 
تابع NWscore
function NWscore(X,Y)
Score(0,0) = 0
for j=1 to length(Y)
Score(0,j) = Score(0,j-1) + Ins(Yj)
for i=1 to length(X)
Score(i,0) = Score(i-1,0) + Del(Xi)
for j=1 to length(Y)
if( Xi == Yj ) scoreSub = Score(i-1,j-1) + Match(Xi, Yj)
else scoreSub = Score(i-1,j-1) + Sub(Xi, Yj)
scoreDel = Score(i-1,j) + Del(Xi)
scoreIns = Score(i,j-1) + Ins(Yj)
Score(i,j) = max(scoreSub, scoreDel, scoreIns)
end
end
for j=0 to length(Y)
LastLine(j) = Score(length(X),j)
return LastLine
این تابع آخرین سطر از ماتریس امتیاز نیدلمن-وانچ را (که معمولاً با F یا score نشان داده میشود) در زمان 

پیادهسازی زیر در MATLAB, با همین پیچیدگی زمانی ولی با حافظهٔ 
function [ lastLine ] = NWscore( X, Y )
% assume length(Y) <= length(X)
scorePrev = zeros(1, length(Y) + 1);
lastLine = zeros(1, length(Y) + 1);
for i = 1:length(Y)
scorePrev(i + 1) = scorePrev(i) + Ins(Y(i));
end
for i = 1:length(X)
lastLine(1) = scorePrev(1) + Del(X(i));
for j = 1:length(Y)
if( X(i) == Y(j) ) s1 = scorePrev(j) + Match(X(i), Y(j));
else s1 = scorePrev(j) + Sub(X(i), Y(j));
end
s2 = scorePrev(j + 1) + Del(X(i));
s3 = lastLine(j) + Ins(Y(j));
lastLine(j + 1) = max([s1, s2, s3]);
end
scorePrev = lastLine;
end
end
تابع Hirschberg
function Hirschberg(X,Y)
Z = ""
W = ""
if length(X) == 0
for i=1 to length(Y)
Z = Z + '-'
W = W + Yi
end
else if length(Y) == 0
for i=1 to length(X)
Z = Z + Xi
W = W + '-'
end
else if length(X) == 1 or length(Y) == 1
(Z,W) = NW(X,Y)
else
xlen = length(X)
xmid = length(X)/2
ylen = length(Y)
ScoreL = NWscore(X1:xmid, Y)
ScoreR = NWscore(rev(Xxmid+1:xlen), rev(Y))
ymid = arg max ScoreL + rev(ScoreR)
(Z,W) = Hirschberg(X1:xmid, y1:ymid) + Hirschberg(Xxmid+1:xlen, Yymid+1:ylen)
end
return (Z,W)
پیادهسازی در MATLAB:
function [ Z, W, maxScore ] = Hirschberg( X, Y )
if( isempty(X) )
Z = char(ones(1, length(Y)) * '-');
W = Y;
elseif( isempty(Y) )
Z = X;
W = char(ones(1, length(X)) * '-');
elseif( length(X) == 1 || length(Y) == 1 )
[Z, W] = NW(X, Y);
else
mid1 = floor(length(X) / 2);
s1 = NWscore(X(1:mid1), Y);
s2 = NWscore(flip(X(mid1 + 1:end)), flip(Y));
[maxScore, mid2] = max(s1 + flip(s2));
mid2 = mid2 - 1;
[temp11, temp21] = Hirschberg(X(1:mid1), Y(1:mid2));
[temp12, temp22] = Hirschberg(X(mid1 + 1:end), Y(mid2 + 1:end));
Z = [temp11, temp12];
W = [temp21, temp22];
end
end
این تابع با استفاده از روش تقسیم و حل، همترازی بهینه و امتیاز این همترازی را بدست میآورد. نحوهٔ پیادهسازی تابع NWscore بیان شد و تابع NW از الگوریتم نیدلمن-وانچ استفاده میکند. زمان اجرای این تابع 

مقایسه با الگوریتم نیدلمن-وانچ
الگوریتم هیرشبرگ، مشابه الگوریتم نیدلمن-وانچ، بهترین امتیاز را با استفاده از برنامهنویسی پویا محاسبه کرده و همترازی متناظر با آن را مییابد.
پیچیدگی زمانی همترازی دو رشته با طولهای m و n در الگوریتم هیرشبرگ 
این الگوریتم تنها 

مثال
فرض کنید دو توالی DNA بنامهای X و Y و امتیازهای همترازسازی به صورت زیر باشند:


در اولین مرحلهٔ الگوریتم هیرشبرگ،

سپس 
C G T A T
0 -2 -4 -6 -8 -10
A -2 -1 -3 -5 -4 -6
C -4 0 -2 -4 -6 -5
G -6 -2 2 0 -2 -4
C -8 -4 0 1 -1 -3
و در نتیجه:
ScoreL = [ -8 -4 0 -2 -1 -3 ]
rev(ScoreR) = [ -3 -1 1 0 -4 -8 ]
Sum = [-11 -5 1 -2 -5 -11]
بیشینهٔ امتیاز در ymid = ۲ است و در نتیجه 
(AGTACGCA,TATGC)
/ \
(AGTA,TA) (CGCA,TGC)
/ \ / \
(AG, ) (TA,TA) (CG,TG) (CA,C)
/ \ / \
(T,T) (A,A) (C,T) (G,G)
برگهای این درخت، همترازی بهینه را نشان میدهند که به صورت زیر است:
W = AGTACGCA
Z = --TATGC-
کاربرد
معمولاً در بیوانفورماتیک از الگوریتم هیرشبرگ برای همترازسازی سراسری توالیهای زیستی (توالیهای آمینواسیدی و توالیهای DNA) استفاده میشود. بلاست (BLAST) و فاستا (FASTA) نمونههای هیوریستیک استفاده از آن هستند.